Découvrez les différentes applications de l’IA en santé – Partie 1
08/02/2024
8 Fév , 2024 read
Modèles de langage pour l’extraction de relations dans les textes cliniques
Perspectives cliniques : Exploiter les modèles de langage pour l’extraction de relations dans les textes cliniques
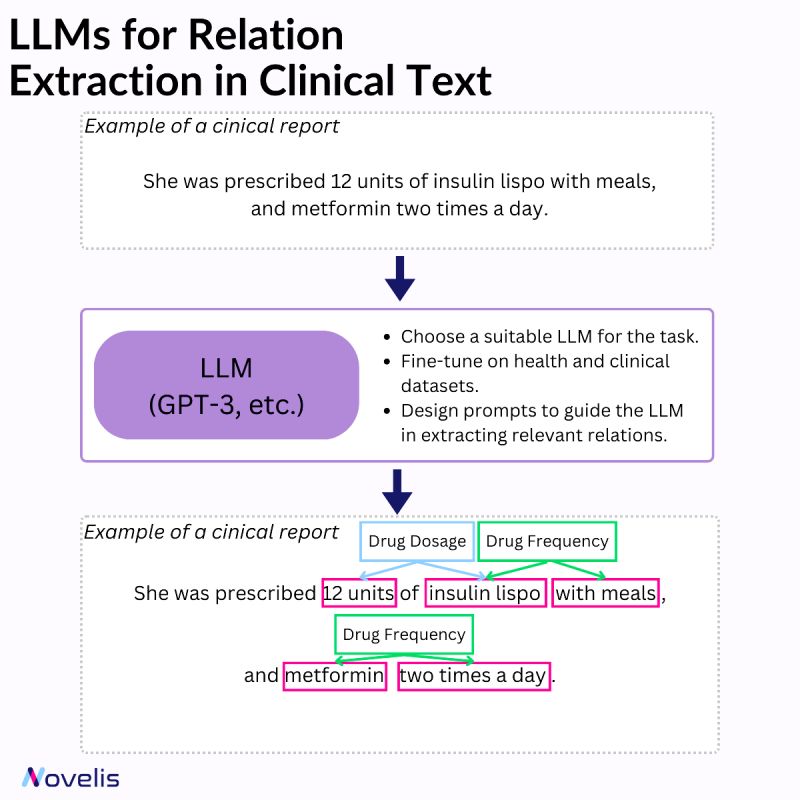
L’extraction de relations consiste à identifier les liens entre des entités nommées dans un texte. Dans le domaine clinique, cela permet de recueillir des informations précieuses sur les maladies, symptômes, traitements et médicaments. Différentes techniques sont utilisées pour la reconnaissance des entités nommées et l’extraction de relations (systèmes basés sur des règles, apprentissage automatique et systèmes hybrides).
Les modèles de langage de grande taille (LLM) ont fortement influencé le domaine de l’apprentissage automatique, en particulier le traitement du langage naturel (NLP). Ces modèles, formés sur d’énormes volumes de données textuelles, peuvent comprendre et générer du texte avec une grande précision. Ils reconnaissent des modèles complexes et des relations sémantiques, gèrent diverses entités et sont adaptables à différents domaines et langues. Lorsqu’ils sont combinés à des heuristiques basées sur des invites et ajustés avec des données cliniques, ils sont particulièrement efficaces pour la reconnaissance des entités nommées et l’extraction des relations.
Pourquoi est-ce essentiel ? Identifier les relations entre différentes entités améliore la compréhension des liens entre les aspects de la santé d’un patient, facilitant ainsi le développement d’interventions efficaces. Par exemple, le soutien à la décision clinique est renforcé par l’extraction de relations entre maladies, symptômes et traitements dans les dossiers médicaux électroniques. De même, identifier les interactions entre différents médicaments contribue à la sécurité des patients et optimise les traitements. L’automatisation de la revue de littérature médicale permet également un accès rapide à des informations pertinentes.
Correspondance entre patients et essais cliniques
Correspondance entre patients et essais cliniques avec des documents enrichis sémantiquement
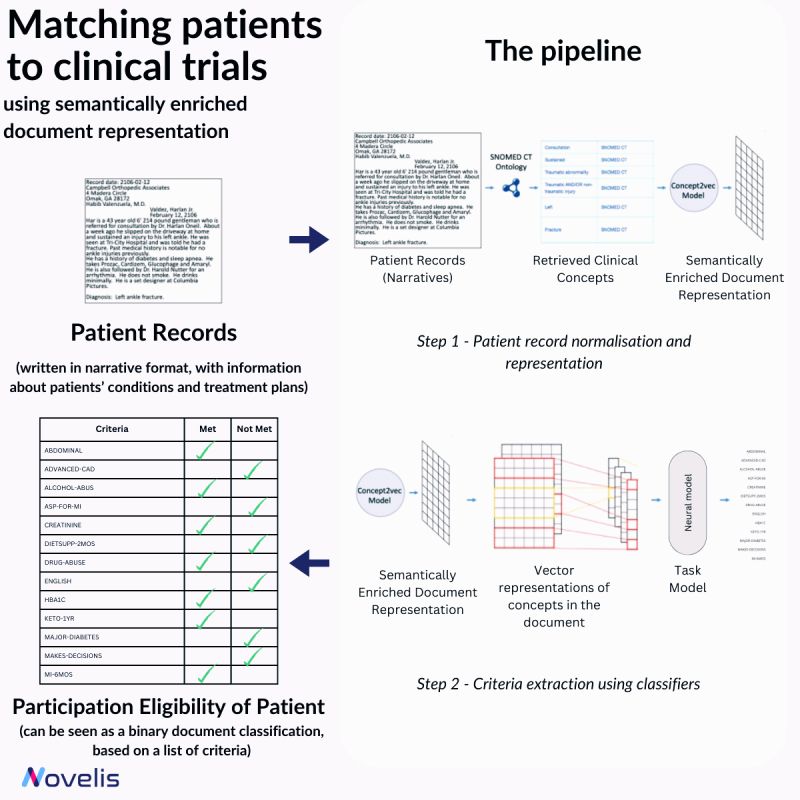
Le recrutement de patients éligibles pour les essais cliniques est essentiel pour le progrès des interventions médicales. Cependant, ce processus manuel est long. Une approche décrite dans « Matching Patients to Clinical Trials Using Semantically Enriched Document Representation » automatise le recrutement en identifiant les patients qui remplissent les critères de sélection à partir de dossiers médicaux.
Cette méthode extrait des informations importantes des documents cliniques, fournit des preuves pour les décisions d’éligibilité basées sur les critères d’inclusion/exclusion et surmonte des défis tels que les variations de style de rapport grâce aux représentations vectorielles sémantiques provenant d’ontologies de domaine. L’ontologie SNOMED CT est utilisée pour normaliser les documents cliniques, tandis que des articles de DBpedia élargissent les concepts en oncologie SNOMED CT. Une méthode neuronale a surpassé les modèles conventionnels avec un score F1 impressionnant de 84 % pour 13 critères d’éligibilité différents.
Pourquoi est-ce essentiel ? Cette recherche est un progrès vers l’amélioration des processus de recrutement pour les essais cliniques, économisant du temps et ouvrant la voie à une recherche et un développement de médicaments plus efficaces.
D’AlphaFold à AlphaMissense
D’AlphaFold à AlphaMissense : modèles pour les variations génétiques
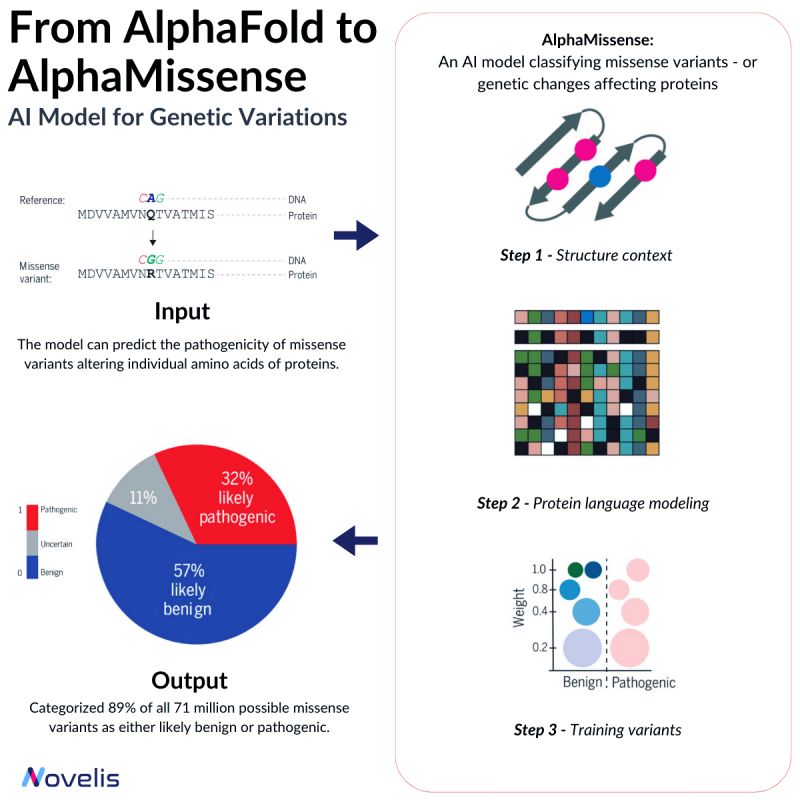
Les mutations faux-sens (missense) sont responsables de nombreuses maladies, telles que le syndrome de Marfan et la maladie de Huntington. Ces mutations provoquent un changement dans la séquence d’acides aminés d’une protéine, pouvant avoir des effets imprévisibles sur l’organisme. Selon leur nature, les mutations faux-sens peuvent être soit pathogènes, soit bénignes. Les variantes pathogènes affectent significativement la fonction des protéines, entraînant des altérations du comportement général de l’organisme, tandis que les variantes bénignes ont peu ou pas d’effet.
Pourquoi est-ce essentiel ? Bien que plus de 4 millions de variantes faux-sens aient été identifiées dans le génome humain, seules environ 2 % d’entre elles ont été classées de manière concluante comme pathogènes ou bénignes. La signification de la majorité de ces variantes reste inconnue, ce qui rend difficile la prédiction de leurs implications cliniques. Par conséquent, des efforts sont en cours pour développer des méthodes efficaces permettant de prédire avec précision les implications cliniques de ces variantes.
Le problème des mutations faux-sens présente des similitudes avec celui du repliement des protéines, les deux cherchant à améliorer l’explicabilité et à prédire les résultats en lien avec les variations de structure des acides aminés. En 2018, DeepMind et l’EMBL-EBI ont lancé AlphaFold, un modèle révolutionnaire pour prédire les structures protéiques, facilitant la prédiction des structures de protéines à partir de séquences d’acides aminés jusque-là inaccessibles.
En tirant parti de l’apprentissage par transfert sur des bases de données publiques annotées (comme BFD, MGnify et UniRef90), DeepMind a développé AlphaMissense, une version améliorée d’AlphaFold qui atteint des prédictions de pointe sur ClinVar (une base de données de mutations génétiques) sans nécessiter un entraînement explicite sur ces données.
L’outil est actuellement disponible sous forme de module complémentaire gratuit dans le logiciel Variant Effect Predictor.
Présentation de GatorTronGPT
Révolutionner la documentation médicale : Présentation de GatorTronGPT

GatorTronGPT est un modèle IA avancé, développé par des chercheurs de l’Université de Floride en collaboration avec NVIDIA. Ce modèle transforme la documentation médicale en permettant de créer des notes précises. Son aptitude à comprendre le langage médical complexe en fait un véritable atout.
Ce modèle de langage a été formé à l’aide de l’architecture GPT-3. Il a été entraîné sur une grande quantité de données, incluant des textes cliniques dépersonnalisés de l’Université de Floride Health et divers textes en anglais issus du jeu de données Pile. GatorTronGPT a ensuite été utilisé pour deux tâches importantes en traitement du langage naturel biomédical : l’extraction de relations biomédicales et le questionnement.
Un test de Turing a été mené pour évaluer la performance de GatorTronGPT. Dans ce test, le modèle a généré des paragraphes de texte clinique synthétique, qui ont été mélangés avec des paragraphes rédigés par des médecins de l’Université de Floride Health. La tâche consistait à identifier les paragraphes écrits par des humains et ceux générés par le modèle, en fonction de la qualité du texte, de la cohérence et de la pertinence. Même des médecins expérimentés n’ont pas pu faire la différence, ce qui démontre la haute qualité des productions de GatorTronGPT.
Propulsé par le cadre GPT-3 d’OpenAI, GatorTronGPT a été entraîné sur le superordinateur HiPerGator, avec le soutien de NVIDIA.
Pourquoi est-ce essentiel ? En reproduisant les compétences rédactionnelles des cliniciens, GatorTronGPT permet aux professionnels de santé de gagner du temps, de réduire l’épuisement professionnel et de se concentrer davantage sur les soins aux patients.
